理化学研究所(理研)放射光科学研究センター生命系放射光利用システム開発チームの上野 剛 専任技師、山本雅貴 チームリーダー、リガク応用技術センター単結晶解析グループの伊藤 翔 研究員の共同研究チームは、深層学習を用いた画像解析により、X線結晶構造解析においてタンパク質結晶試料を自動的に検出するプログラム「DeepCentering」を開発した。本研究成果は、タンパク質結晶の自動位置決めを実現し、全自動データ収集、さらには自動構造解析への応用が期待できる。
本研究は、英国の科学雑誌『Journal of Synchrotron Radiation』7月1日号と、6月3日付けオンライン版に掲載された。
タンパク質の構造情報は、そのタンパク質の機能を理解する上で非常に重要で、とりわけ、放射光施設における高輝度X線を利用した結晶構造解析は、これまで多くのタンパク質の構造を明らかにし、その機能を理解するために重要な役割を果たしてきた。
X線結晶構造解析では、ゴニオメーター上にマウントされた結晶をどの方向に回してもX線の光路上に位置させる作業(結晶センタリング)が必要となる。従来、この作業はビームライン利用者が目視で行うか、強度を弱めたX線を結晶に照射することで結晶の位置を検出して行うのが主流だった。
今回、共同研究チームは、結晶センタリングを自動化するために、深層学習を用いた画像処理技術を応用することを試みた。
共同研究チームは、深層学習を用いて、クライオループおよび結晶を認識するプログラム「DeepCentering」を開発。この際、クライオループを認識するための教師データとしては、これまでに大型放射光施設「SPring-8」のタンパク質結晶構造解析ビームライン(BL26B2)で取得した約6000枚のクライオループ画像を利用した。次に、結晶を認識するための教師データとしては、実際のタンパク質結晶画像では外形があいまいな例が数多く含まれるため、単純な多角形の画像を約400枚自動生成して利用した。その結果、学習が効率良く進み、精度の高い検出結果を得ることに成功したもの(図1)。
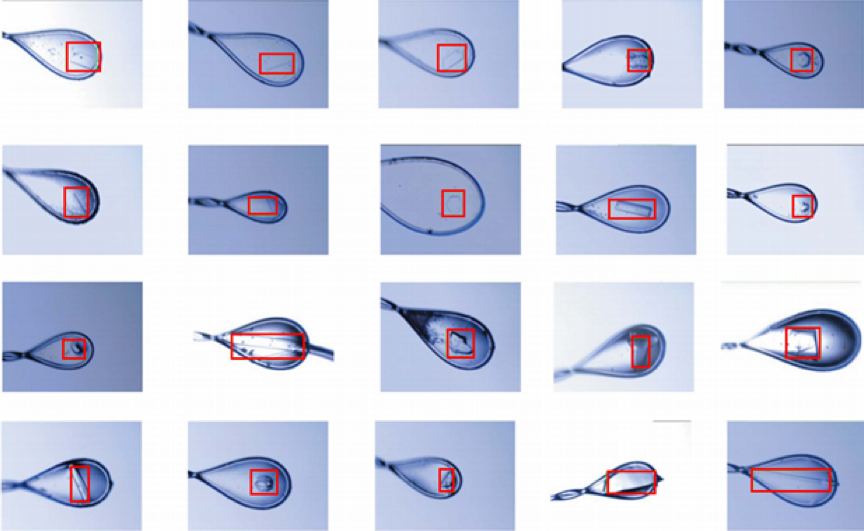
ph1を使用:DeepCenteringによる結晶検出:タンパク質結晶をマウントしたクライオループ。赤色の長方形で囲まれている箇所が結晶と認識された範囲。クライオループの大きさは、約200~600µm
これまでも画像処理技術を用いた結晶検出プログラムは開発されてきたが、試料観察カメラのコントラストなどが変わると検出精度が低下するなどの問題があった。DeepCenteringでは、そのような場合でも正確にクライオループや結晶を検出し、これまで開発されてきた画像処理ベースでの結晶検出プログラムの問題を解決。DeepCenteringと、すでに研究チームが開発しているビームライン制御ソフトウェア、回折データ自動処理プログラム、および自動構造解析プログラムを用いて、データ取得および構造解析の自動化に成功した。
また、DeepCenteringの機能の一部を利用して、自動センタリングした試料に対し、目視によるセンタリング結果との画像比較による自動位置修正を行うことで、より高精度な結晶位置決めも可能となる。この機能は、ビームラインでのメールインデータ収集[7]に利用されている。
DeepCenteringの開発により、X線照射を必要とすることなく試料検出の自動化が可能となった。これにより、特に放射線損傷の影響を受けやすい試料や室温での自動回折データ収集での活用が期待できる。
また、ビームラインでのデータ収集と構造解析の全自動化により、特定のタンパク質と化合物の複合体構造解析を大量に実施する化合物スクリーニングや、既に類似構造が明らかになっているタンパク質の結晶化条件を探索する結晶回折スクリーニングや構造解析など、大量の結晶試料を取り扱う実験の効率化が推進されると見られている。

